組蛋白修飾如何影響基因表達?
日期:2024-02-06 11:37:30
正如題為 "DNA壓縮的強大偽裝--組蛋白 "的文章所述,組蛋白修飾是對組蛋白進行的一系列翻譯后修飾(PTM),包括甲基化、磷酸化、乙酰化、泛素化和蘇木酰化。對組蛋白進行的 PTM 可通過改變染色質結構或招募組蛋白修飾因子來影響基因表達。但其機制仍不清楚。
在真核細胞的細胞核中,DNA纏繞在組蛋白的八聚體上形成核小體,其結構就像 "串珠",是染色質的基本單位。染色質進一步折疊成更高層次的結構,或松或緊,這有助于決定DNA的可及性。
兩位科學家因其發現獲得了2018年阿爾伯特-拉斯克基礎醫學研究獎,他們的發現闡明了基因表達如何受到組蛋白化學修飾的影響,組蛋白是染色體中包裝DNA的蛋白質。
美國加州大學洛杉磯分校教授邁克爾-格倫斯坦(Michael Grunstein)通過在酵母中進行艱苦卓絕的遺傳研究,證明了組蛋白對活細胞內基因活動的巨大影響,并為理解特定氨基酸在這一過程中的關鍵作用奠定了基礎 [1]。洛克菲勒大學教授 C. David Allis 發現了一種能將特定化學基團連接到組蛋白中特定氨基酸上的酶,這種組蛋白修飾酶被證明是一種成熟的基因共激活因子,其生化功能一直困擾著研究人員 [2]。格倫斯坦和阿里斯揭開了基因控制的一個隱藏層次,開辟了一個新領域。
這里,我們以組蛋白乙酰化為例。如圖 1 所示。通過組蛋白乙酰化激活轉錄,帶有正電荷的乙酰賴氨酸可以使組蛋白與帶有負電荷的 DNA 緊密結合。因此,轉錄機制無法訪問 DNA,基因也就失去了活性。在組蛋白尾部的特定賴氨酸上添加乙酰基可以中和正電荷,松開核小體對 DNA 的控制。這一過程可使轉錄機制進入 DNA,從而使基因活躍起來。其他修飾以不同方式影響轉錄 [3] [4]。
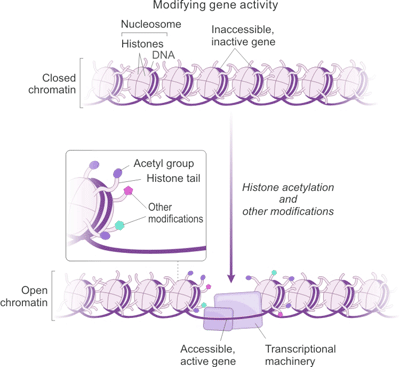
圖1. 組蛋白乙酰化與基因表達的相關性
綜上所述,對于組蛋白修飾與基因表達之間的相關性,有句話可以很好地描述這種關系:"轉錄調控是一個復雜而動態的過程,組蛋白修飾是其中的關鍵組成部分"。有些組蛋白修飾是活躍轉錄的結果;然而,這些組蛋白修飾提供了對近期轉錄活動的記憶,并為額外的轉錄調控提供了信號。許多組蛋白修飾與活躍轉錄無關,但在轉錄調控過程中發揮作用。全基因組實驗、生物信息學分析以及可將表觀遺傳酶靶向特定 DNA 序列的新技術的快速發展,將有助于闡明無數組蛋白修飾的成因和生物學功能 [5]。
參考文獻:
[1] Shahbazian, M.D., and Grunstein, M. Functions of site-specific histone acetylation and deacetylation [J]. Annu. Rev. Biochem. 2007, 76, 75-100.
[2] Lewis, P.W., Mueller, M.M., et al. Inhibition of PRC2 activity by gain-of-function mutations in pediatric gliobastoma [J]. Science. 2013, 340, 857-861.
[3] Kurdistani, S.K., Tavazoie, S, et al. Mapping global histone acetylation patterns to gene expression [J]. Cell. 2004, 117, 721-733.
[4] Maze, I., Noh, K.M., et al. Every amino acid matters: essential contributions of histone variants to mammalian development and disease [J]. Nat. Rev. Genet. 2014, 15, 259-271.
[5] Xianjun Dong and Zhiping Weng. The correlation between histone modifications and gene expression [J]. Epigenomics. 2013 Apr; 5(2): 113–116.
上一篇: 組蛋白修飾:基因調控和疾病發病機制的意義
下一篇: 組蛋白修飾與癌癥












